Métodos comparativos filogenéticos
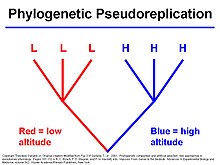
Los métodos comparativos filogenéticos (PCM) utilizan información sobre las relaciones históricas de los linajes (filogenias) para probar hipótesis evolutivas. El método comparativo tiene una larga historia en biología evolutiva; de hecho, Charles Darwin utilizó las diferencias y similitudes entre las especies como una fuente importante de evidencia en El origen de las especies. Sin embargo, el hecho de que los linajes estrechamente relacionados compartan muchos rasgos y combinaciones de rasgos como resultado del proceso de descenso con modificación significa que los linajes no son independientes. Esta realización inspiró el desarrollo de métodos comparativos explícitamente filogenéticos. Inicialmente, estos métodos se desarrollaron principalmente para controlar la historia filogenética al probar la adaptación; sin embargo, en los últimos años, el uso del término se ha ampliado para incluir cualquier uso de las filogenias en las pruebas estadísticas. Aunque la mayoría de los estudios que emplean PCM se centran en organismos existentes; muchos métodos también pueden aplicarse a taxones extintos y pueden incorporar información del registro fósil.
Los PCM generalmente se pueden dividir en dos tipos de enfoques: aquellos que infieren la historia evolutiva de algunos caracteres (fenotípicos o genéticos) a través de una filogenia y aquellos que infieren el proceso de ramificación evolutiva (tasas de diversificación), aunque hay algunos enfoques que sí hacen ambos a la vez. Por lo general, el árbol que se usa junto con los PCM se ha estimado de forma independiente, de modo que se supone que se conocen las relaciones entre los linajes y la longitud de las ramas que los separan.
Aplicaciones
Los enfoques comparativos filogenéticos pueden complementar otras formas de estudiar la adaptación, como el estudio de poblaciones naturales, estudios experimentales y modelos matemáticos. Hacer comparaciones interespecíficas permite a los investigadores evaluar la generalidad de los fenómenos evolutivos considerando eventos evolutivos independientes. Este enfoque es particularmente útil cuando hay poca o ninguna variación dentro de las especies. Y debido a que se pueden usar para modelar explícitamente los procesos evolutivos que ocurren durante períodos de tiempo muy largos, pueden proporcionar información sobre las cuestiones macroevolutivas, que antes eran el dominio exclusivo de la paleontología.

Los métodos comparativos filogenéticos se aplican comúnmente a preguntas tales como:
- ¿Cuál es la pendiente de una relación de escala alométrica?
→ Ejemplo: ¿cómo varía la masa cerebral en relación con la masa corporal?
- ¿Diferentes clados de organismos difieren con respecto a algún rasgo fenotípico?
→ Ejemplo: ¿los cánidos tienen corazones más grandes que los felinos?
- ¿Los grupos de especies que comparten una característica de comportamiento o ecológica (p. ej., sistema social, dieta) difieren en el fenotipo promedio?
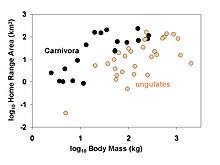
→ Ejemplo: ¿Los carnívoros tienen rangos de hogar más grandes que los herbívoros?
→ Ejemplo: ¿dónde evolucionó la endotermia en el linaje que llevó a los mamíferos?
→ Ejemplo: ¿dónde, cuándo y por qué evolucionaron las placentas y la viviparidad?
- ¿Un rasgo muestra una señal filogenética significativa en un grupo particular de organismos? ¿Ciertos tipos de rasgos tienden a "seguir la filogenia" más que otros?
→ Ejemplo: ¿son los rasgos de comportamiento más lábiles durante la evolución?
- ¿Las diferencias de especies en las características de la historia de la vida se compensan, como en el llamado continuo lento-lento?
→ Ejemplo: ¿por qué las especies de cuerpo pequeño tienen vidas más cortas que sus parientes más grandes?
Contrastes filogenéticamente independientes
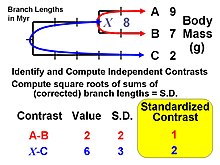
Felsenstein propuso el primer método estadístico general en 1985 para incorporar información filogenética, es decir, el primero que podría utilizar cualquier topología arbitraria (orden de ramificación) y un conjunto específico de longitudes de rama. El método ahora se reconoce como un algoritmo que implementa un caso especial de lo que se denominan modelos filogenéticos de mínimos cuadrados generalizados. La lógica del método es utilizar información filogenética (y un supuesto movimiento browniano como modelo de evolución de rasgos) para transformar los datos originales de las puntas (valores medios para un conjunto de especies) en valores que son estadísticamente independientes y distribuidos de forma idéntica.
El algoritmo implica calcular valores en nodos internos como un paso intermedio, pero generalmente no se usan para inferencias por sí mismos. Se produce una excepción para el nodo basal (raíz), que se puede interpretar como una estimación del valor ancestral para todo el árbol (suponiendo que no se hayan producido tendencias evolutivas direccionales [p. ej., La regla de Cope ]) o como una estimación ponderada filogenéticamente de media para todo el conjunto de especies de puntas (taxones terminales). El valor en la raíz es equivalente al obtenido del algoritmo de "parsimonia de cambio cuadrado" y también es la estimación de máxima verosimilitud bajo el movimiento browniano. El álgebra de contrastes independiente también se puede usar para calcular un error estándar o un intervalo de confianza .
Mínimos cuadrados filogenéticos (PGLS)
Probablemente el PCM más utilizado es el de los mínimos cuadrados filogenéticos (PGLS). Este enfoque se utiliza para comprobar si existe una relación entre dos (o más) variables, al tiempo que se tiene en cuenta el hecho de que el linaje no es independiente. El método es un caso especial de mínimos cuadrados generalizados (GLS) y, como tal, el estimador PGLS también es imparcial, consistente, eficiente y asintóticamente normal. En muchas situaciones estadísticas donde se usa GLS (o, mínimos cuadrados ordinarios [OLS]), se asume que los errores residuales ε son variables aleatorias independientes e idénticamente distribuidas que se asume que son normales
mientras que en PGLS se asume que los errores se distribuyen como
donde V es una matriz de varianza esperada y covarianza de los residuos dado un modelo evolutivo y un árbol filogenético. Por lo tanto, es la estructura de los residuos y no las propias variables las que muestran la señal filogenética. Esto ha sido durante mucho tiempo una fuente de confusión en la literatura científica. Se han propuesto varios modelos para la estructura de V, como el movimiento browniano Ornstein-Uhlenbeck, y el modelo λ de Pagel. (Cuando se usa un modelo de movimiento browniano, PGLS es idéntico al estimador de contrastes independiente). En PGLS, los parámetros del modelo evolutivo se suelen estimar conjuntamente con los parámetros de regresión.
PGLS solo se puede aplicar a preguntas donde la variable dependiente se distribuye continuamente; sin embargo, el árbol filogenético también puede incorporarse en la distribución residual de modelos lineales generalizados, lo que hace posible generalizar el enfoque de un conjunto más amplio de distribuciones para la respuesta.
Simulaciones informáticas de Monte Carlo
Martins y Garland propusieron en 1991 que una forma de explicar las relaciones filogenéticas al realizar análisis estadísticos era usar simulaciones por computadora para crear muchos conjuntos de datos que fueran consistentes con la hipótesis nula bajo prueba (por ejemplo, no hay correlación entre dos rasgos, no diferencia entre dos grupos de especies definidas ecológicamente) pero que imitan la evolución a lo largo del árbol filogenético relevante. Si dichos conjuntos de datos (típicamente 1,000 o más) se analizan con el mismo procedimiento estadístico que se usa para analizar un conjunto de datos real, entonces los resultados de los conjuntos de datos simulados se pueden usar para crear la filogenética correcta (o "PC" ) distribuciones nulas del estadístico de prueba (por ejemplo, un coeficiente de correlación, t, F). Tales enfoques de simulación también se pueden combinar con métodos como los contrastes filogenéticamente independientes o PGLS.
Véase también
- Alometría
- Ecología del comportamiento
- Biodiversidad
- Bioinformática
- Cladistics
- Método comparativo (lingüística)
- Fisiología comparada
- Filogenética computacional
- Método de cobertura del disco
- Ecofisiología
- Neurobiología evolutiva
- Fisiología evolutiva
- Mínimos cuadrados generalizados (GLS)
- Modelo lineal generalizado
- Máxima verosimilitud
- Máxima parsimonia
- Selección sexual
- Sistemática
Otras lecturas
- Ackerly, D. D. 1999. Comparative plant ecology and the role of phylogenetic information. Pages 391–413 in M. C. Press, J. D. Scholes, and M. G. Braker, eds. Physiological plant ecology. The 39th symposium of the British Ecological Society held at the University of York 7–9 September 1998. Blackwell Science, Oxford, U.K.
- Berenbrink, M.; Koldkjær, P.; Kepp, O.; Cossins, A. R. (2005). «Evolution of oxygen secretion in fishes and the emergence of a complex physiological system». Science 307: 1752-1757. PMID 15774753. doi:10.1126/science.1107793.
- Blomberg, S. P.; Jr; Garland, T.; Ives, A. R. (2003). «Testing for phylogenetic signal in comparative data: behavioral traits are more labile» (PDF). Evolution 57: 717-745. doi:10.1554/0014-3820(2003)057[0717:tfpsic]2.0.co;2.
- Brooks, D. R., and D. A. McLennan. 1991. Phylogeny, ecology, and behavior: a research program in comparative biology. Univ. Chicago Press, Chicago. 434 pp.
- Cheverud, J. M.; Dow, M. M.; Leutenegger, W. (1985). «The quantitative assessment of phylogenetic constraints in comparative analyses: sexual dimorphism in body weight among primates». Evolution 39: 1335-1351. doi:10.2307/2408790.
- Eggleton, P., and R. I. Vane-Wright, eds. 1994. Phylogenetics and ecology. Linnean Society Symposium Series Number 17. Academic Press, London.
- Felsenstein, J. 2004. Inferring phylogenies. Sinauer Associates, Sunderland, Mass. xx + 664 pp.
- Freckleton, R. P.; Harvey, P. H.; Pagel, M. (2002). «Phylogenetic analysis and comparative data: a test and review of evidence». American Naturalist 160: 712-726. PMID 18707460. doi:10.1086/343873.
- Garland Jr, T; Ives, A. R. (2000). «Using the past to predict the present: Confidence intervals for regression equations in phylogenetic comparative methods» (PDF). American Naturalist 155: 346-364. doi:10.1086/303327.
- Garland Jr, T.; Bennett, A. F.; Rezende, E. L. (2005). «Phylogenetic approaches in comparative physiology» (PDF). Journal of Experimental Biology 208: 3015-3035. PMID 16081601. doi:10.1242/jeb.01745.
- Garland Jr, T.; Harvey, P. H.; Ives, A. R. (1992). «Procedures for the analysis of comparative data using phylogenetically independent contrasts» (PDF). Systematic Biology 41: 18-32. doi:10.2307/2992503.
- Gittleman, J. L.; Kot, M. (1990). «Adaptation: statistics and a null model for estimating phylogenetic effects». Systematic Zoology 39: 227-241. doi:10.2307/2992183.
- Hadfield, J. D; Nakagawa, S. (2010). «General quantitative genetic methods for comparative biology: phylogenies, taxonomies and multi-trait models for continuous and categorical characters». Journal of Evolutionary Biology 23: 494-508. PMID 20070460. doi:10.1111/j.1420-9101.2009.01915.x.
- Herrada, E. A.; Tessone, C. J.; Klemm, K.; Eguiluz, V. M.; Hernandez-Garcia, E.; Duarte, C. M. (2008). «Universal Scaling in the Branching of the Tree of Life». PLOS ONE 3 (7): e2757. doi:10.1371/journal.pone.0002757.
- Housworth, E. A.; Martins, E. P.; Lynch, M. (2004). «The phylogenetic mixed model» (PDF). American Naturalist 163: 84-96. doi:10.1086/380570.
- Ives, A. R. 2018. Mixed and phylogenetic models: a conceptual introduction to correlated data. leanpub.com, 125 pp., https://leanpub.com/correlateddata
- Ives, A. R.; Midford, P. E.; Garland Jr, T. (2007). «Within-species variation and measurement error in phylogenetic comparative methods». Systematic Biology 56: 252-270. doi:10.1080/10635150701313830.
- Maddison, D. R. (1994). «Phylogenetic methods for inferring the evolutionary history and process of change in discretely valued characters». Annual Review of Entomology 39: 267-292. doi:10.1146/annurev.ento.39.1.267.
- Maddison, W. P. (1990). «A method for testing the correlated evolution of two binary characters: Are gains or losses concentrated on certain branches of a phylogenetic tree?». Evolution 44: 539-557. doi:10.2307/2409434.
- Maddison, W. P., and D. R. Maddison. 1992. MacClade. Analysis of phylogeny and character evolution. Version 3. Sinauer Associates, Sunderland, Mass. 398 pp.
- Martins, E. P., ed. 1996. Phylogenies and the comparative method in animal behavior. Oxford University Press, Oxford. 415 pp.
- Martins, E. P.; Hansen, T. F. (1997). «Phylogenies and the comparative method: a general approach to incorporating phylogenetic information into the analysis of interspecific data». American Naturalist 149: 646-667. doi:10.1086/286013. Erratum Am. Nat. 153:448.
- Nunn, C. L.; Barton, R. A. (2001). «Comparative methods for studying primate adaptation and allometry». Evolutionary Anthropology 10: 81-98. doi:10.1002/evan.1019.
- Oakley, T. H.; Gu, Z.; Abouheif, E.; Patel, N. H.; Li, W.-H. (2005). «Comparative methods for the analysis of gene-expression evolution: an example using yeast functional genomic data» (PDF). Molecular Biology and Evolution 22: 40-50. doi:10.1093/molbev/msh257.
- O'Meara, B. C.; Ané, C. M.; Sanderson, M. J.; Wainwright, P. C. (2006). «Testing for different rates of continuous trait evolution in different groups using likelihood» (PDF). Evolution 60: 922-933. doi:10.1554/05-130.1.
- Organ, C. L.; Shedlock, A. M.; Meade, A.; Pagel, M.; Edwards, S. V. (2007). «Origin of avian genome size and structure in non-avian dinosaurs». Nature 446: 180-184. PMID 17344851. doi:10.1038/nature05621.
- Page, R. D. M., ed. 2003. Tangled trees: phylogeny, cospeciation, and coevolution. University of Chicago Press, Chicago.
- Pagel, M. D. (1993). «Seeking the evolutionary regression coefficient: an analysis of what comparative methods measure». Journal of Theoretical Biology 164: 191-205. doi:10.1006/jtbi.1993.1148.
- Pagel, M (1999). «Inferring the historical patterns of biological evolution». Nature 401: 877-884.
- Paradis, E (2005). «Statistical analysis of diversification with species traits». Evolution 59: 1-12. doi:10.1554/04-231.
- Paradis, E.; Claude, J. (2002). «Analysis of comparative data using generalized estimating equations». Journal of Theoretical Biology 218: 175-185. doi:10.1006/jtbi.2002.3066.
- Purvis, A.; Garland Jr, T. (1993). «Polytomies in comparative analyses of continuous characters» (PDF). Systematic Biology 42: 569-575. doi:10.2307/2992489.
- Rezende, E. L., and T. Garland, Jr. 2003. Comparaciones interespecíficas y métodos estadísticos filogenéticos. Pages 79–98 in F. Bozinovic, ed. Fisiología Ecológica & Evolutiva. Teoría y casos de estudios en animales. Ediciones Universidad Católica de Chile, Santiago. PDF
- Rezende, E.L.; Diniz-Filho, J.A.F (2012). «Phylogenetic analyses: comparing species to infer adaptations and physiological mechanisms» (PDF). Comprehensive Physiology 2: 639-674. (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).
- Ridley, M. 1983. The explanation of organic diversity: The comparative method and adaptations for mating. Clarendon, Oxford, U.K.
- Rohlf, F. J. (2001). «Comparative methods for the analysis of continuous variables: geometric interpretations». Evolution 55: 2143-2160. doi:10.1111/j.0014-3820.2001.tb00731.x.
- Rohlf, F. J. (2006). «A comment on phylogenetic correction». Evolution 60: 1509-1515. doi:10.1554/05-550.1.
- Sanford, G. M.; Lutterschmidt, W. I.; Hutchison, V. H. (2002). «The comparative method revisited». BioScience 52: 830-836. doi:10.1641/0006-3568(2002)052[0830:tcmr]2.0.co;2.
- Schluter, D.; Price, T.; Mooers, A. O.; Ludwig, D. (1997). «Likelihood of ancestor states in adaptive radiation». Evolution 51: 1699-1711. doi:10.2307/2410994.
- Smith, R. J.; Cheverud, J. M. (2002). «Scaling of sexual size dimorphism in body mass: a phylogenetic analysis of Rensch's rule in primates». International Journal of Primatology 23: 1095-1135. doi:10.1023/A:1019654100876.
- Steppan, S. J.; Phillips, P. C.; Houle, D. (2002). «Comparative quantitative genetics: evolution of the G matrix» (PDF). Trends in Ecology and Evolution 17: 320-327. doi:10.1016/s0169-5347(02)02505-3.
- Vanhooydonck, B.; Van Damme, R. (1999). «Evolutionary relationships between body shape and habitat use in lacertid lizards». Evolutionary Ecology Research 1: 785-805.
Enlaces externos
- Adaptación y el método comparativo en línea, con ejemplos resueltos de contrastes filogenéticamente independientes y prueba de dominio.
- Listado de programas de filogenia.
- Phylocomm
- Herramientas filogenéticas para biología comparativa
- Sitio web de la filogenia del sueño
- Árbol de la vida
Revistas
- Naturalista americano
- Ecología del comportamiento
- Ecología
- Evolución
- Investigación en ecología evolutiva
- Ecología funcional
- Revista de biología evolutiva
- Transacciones filosóficas de la Royal Society de Londres B
- Zoología fisiológica y bioquímica.
- Biología sistemática
Paquetes de software (lista incompleta)
- Análisis de la filogenética y la evolución.
- BayesTraits
- Análisis comparativo por contrastes independientes
- COMPARAR
- Lista de Felsenstein
- Mesquite PDAP: PDTree para Mesquite
- mvmorfo
- ouch: Ornstein-Uhlenbeck para Hipótesis Comparativas
- PDAP: Programas de Análisis de Diversidad Fenotípica
- Phylocom
- Regresión filogenética
- PHYSIG
Laboratorios
- Ackerly
- Bininda-Emonds
- Blomberg
- Butler
- Felsenstein
- Freckleton
- Garland
- Gittleman
- Grafen
- Hansen
- Harmon
- Harvey
- Housworth
- Irschick
- Ives
- Losos
- Martins
- Mooers
- Mort
- Nunn
- Oakley
- Page
- Pagel
- Paradis
- Purvis
- Rambaut
- Rohlf
- Sanderson
| Control de autoridades |
|
|---|
-
 Datos: Q7189434
Datos: Q7189434